Imaging of Matter
Elektronenbeugung zeigt winzige Kristalle in neuem Licht
26. Februar 2020
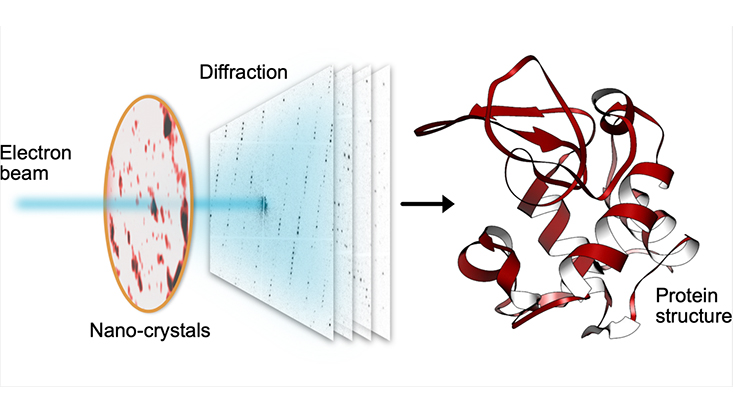
Foto: Robert Bücker
Um die biologischen Funktionen von Proteinen zu verstehen, ist es unerlässlich, ihre Struktur zu erforschen. Dank ihrer winzigen Dimensionen und Zerbrechlichkeit sind diese Strukturen jedoch schwer zu bestimmen. Die benötigte immense Dosis an hochenergetischer Röntgenstrahlung richtet in den zu untersuchenden Proteinen große Schäden und verhindert oft die Strukturauflösung. Nun haben Forscher am Max-Planck-Institut für Struktur und Dynamik der Materie (MPSD) und am Deutschen Elektronensychrotron (DESY) in Hamburg eine innovative neue Methode entwickelt, die diese Probleme umgeht und weit verbreitete, kosteneffektive Technologien nutzt. Die Beschreibung dieser Methode wurde im Fachblatt "Nature Communications" veröffentlicht.
Seit Jahrzehnten suchen Forschende aus diversen Feldern, von der Physik bis zur Biologie und Biochemie, nach kreativen Lösungen für das Strahlungsschäden-Problem. Ein neuer Ansatz ist die Nutzung von ultrakurzen und intensiven Röntgenblitzen an Großinstrumenten wie dem XFEL in Hamburg, welches hochaufgelöste Abbildungen produziert, bevor die Proteine wortwörtlich explodieren. Diese Methode besticht durch spektakuläre Ergebnisse, allerdings benötigt man große und teure Teilchenbeschleuniger, um Röntgenstrahlen der nötigen Brillianz zu erzeugen. Eine andere Methode setzt stattdessen Elektronenstrahlen ein, die sanfter mit den zarten Biomolekülen umgehen und einfacher zu erzeugen sind. Diese kommt zum Beispiel am Center for Structural Systems Biology (CSSB) in Hamburg zum Einsatz.
Big Data-Analysen und neueste Kameratechnologien
Das Team von DESY und dem MPSD am Center for Free-Electron Laser Science (CFEL), zu dem auch Forschende des Exzellenzclusters "CUI: Advanced Imaging of Matter" gehören, hat nun auf raffinierte Weise diese Ansätze mit Big Data-Analysen und den neuesten Kameratechnologien kombiniert und dadurch hochaufgelöste Proteinstrukturen aus relativ leicht erhältlichen Nanokristallen gewonnen. Hierzu entwickelten die Wissenschaftlerinnen und Wissenschaftler die serielle Elektronenbeugung (Serial Electron Diffraction), die experimentelle Methoden aus der Röntgenkristallographie adaptiert, um Beugungsmuster aus tausenden Kristallen zu gewinnen und diese zu verarbeiten. Sie verteilten diese Kristalle einfach auf einem dünnen Kohlefilm und legten sie in ein Elektronenmikroskop, wie es in vielen Laboren vorhanden ist. Der Elektronenstrahl wird dazu gebracht, von einem Nanokristall zum nächsten zu hüpfen, um Beugungsdaten zu sammeln. Die Nutzung von Nanokristallen reduziert nicht nur die benötigte Menge der oft seltenen und teuren Proben, sondern erspart den Forschenden zudem das Züchten großer Proteinkristalle, wie bei konventionellen Röntgenmethoden – oftmals eine extrem schwierige Aufgabe.
"Diffraction during Destruction-Movie"
Um die vom Elektronenstrahl verursachten Schäden zu umgehen, wird statt eines Einzelbildes ein kurzer Film mit einer Hochgeschwindigkeitskamera gedreht, während der Elektronenstrahl auf einem Kristall ruht. Der Film zeigt, wie der Kristall buchstäblich „dahinschmilzt“ und dennoch bleiben ausreichende Informationen im "Diffraction during Destruction-Movie", um die Daten so zu rekonstruieren, als hätte es beinahe keinen Schaden gegeben. Dieser Prozess wird innerhalb weniger Stunden bei tausenden von Nanokristallen wiederholt, dann werden die enormen Datenmengen mithilfe am DESY entwickelter, spezieller Software in eine hochaufgelöste Proteinstruktur umgewandelt.
Außer bei Proteinen und anderen Biomolekülen kann die serielle Elektronenbeugung auch für viele neuartige Funktionsmaterialien eingesetzt werden – wie zum Beispiel Perovskite und metallorganische Frameworks, die vielversprechende Kandidaten für zukünftige Anwendungen in Solarzellen und Wasserstoffspeichern darstellen.
Das Forschungsteam ist begeistert über die Einfachheit dieser Methode mit ihren verhältnismäßig geringen Anforderungen an wertvolle Proben und aufwändige Laborausstattung und erhofft sich ihre Verbreitung vom MPSD in Laboren weltweit. Text: MPSD, red.
Originalpublikation:
R. Bücker, P. Hogan-Lamarre, P. Mehrabi, E.-C. Schulz, L. Bultema, Y. Gevorkov, W. Brehm, O. Yefanov, D. Oberthür, G. Kassier, R. J. D. Miller
"Serial protein crystallography in an electron microscope"
Nature Communications 11, 996 (2020)


